Starnberg, 11. Jan. 2019 - Vom Forschungslabor in die Praxis: Catalog arbeitet mit Cambridge Consultants an DNA-Datenspeicher als HDD- und Tape-Alternative...
Um was es hier geht: Um eine ziemlich steile These! Die Datenspeicherung "kann auch biologisch"... zumindest im Bereich der Langzeitdatenspeicherung von sehr großen Datenbeständen. Wissenschaftler forschen bereits seit längerem an verschiedenen Möglichkeiten, DNA-Material als Speicherbaustein zu verwenden (1), aber es wurde nicht unbedingt erwartet, dass erste Ergebnisse bereits auf überschaubare Sicht (ca. 10 Jahre) kommerziell auch nutzbar sein könnten (siehe z.B. Clelland, C.T. et al. 1999. Hiding messages in DNA microdots. Nature. 399, (1999), 533–534). Ein Grund: Die bislang erforschten Verfahren sind langsam, skalieren nicht und zudem sehr teuer.
Anders hingegen die von CATALOG Infinite Data Archives- einem US-Startup (1) - entwickelte Methode, welche nach Angaben der Forscher einen neuen Ansatz zur DNA-Speicherung für Archivzwecke verfolgt (keine neue Synthese notwendig / weitere Details finden Sie im Text unten). Die Motivation dahinter: bereits um 2025 sollen laut Catalog konventionelle Speichermedien wie Festplatten in Rechenzentren, die als Basis für Cloud Services genutzt werden, nicht mehr in der Lage sein, den Kapazitätsbedarf für die Datenspeicherung zu wirtschaftlich vertretbaren Kosten zu decken. DNA-Datenspeicherung, die synthetische DNA verwendet, ist hingegen platzsparend, hochgradig nachhaltig und kann dabei enorme Datenmengen auf kleinstem Raum speichern.
Die Verwendung von DNA zur Archivierung von „cold data“ - dem idealen Anwendungsfall für DNA-Storage - scheint vielversprechend, da sie mit einer ungefähren Grenze von 1 Exabyte/mm³ (10⁹ GB/mm³) hochdicht und langlebig ist sowie eine Halbwertszeit von rund 500 Jahren aufweist. Im Vergleich dazu liegt die durchschnittliche Haltbarkeit von Flash und Festplatte deutlich darunter (für Tape Media sind bis 30 Jahre spezifiziert). Bisherige Langzeitarchivierungslösungen erfordern zudem regelmäßige HW- und SW-Updates, um beschädigte Daten zu bereinigen, fehlerhafte Komponenten zu ersetzen und den gesamten Technologie-Stack insgesamt auf dem neuesten Stand zu halten (refresh, data migration).
Zusammen mit dem UK-Innovationsspezialisten Cambridge Consultants* hat Catalog im Rahmen der SynBioBeta 2018 im Oktober letzten Jahres in San Francisco nun eine Partnerschaft mit Plänen zur Entwicklung und dem Bau eine Maschine vorgestellt, um Speicherdaten in synthetische DNA zu kodieren. Die Maschine soll den Kodierungsprozess skalieren (bislang nicht möglich), so dass DNA-Storage erschwinglich und auch schnell genug für erste professionelle Anwendungen sein soll. Eine erste Lösung ist demnach noch für 2019 geplant. Mit dieser Technologie könnten laut Entwickler künftig die Informationsinhalte ganzer Rechenzentren in etwa eine Handfläche passen.
HIntergrund: Das DNA Speicherprinzip*
Die Grundeinheit der DNA-Speicherung ist ein DNA-Strang, der etwa 100-200 Nukleotide lang ist und 50-100 Bit insgesamt speichern kann. Daher wird ein Datenobjekt auf eine sehr große Anzahl von DNA-Strängen abgebildet. Die DNA-Stränge werden in "Pools" gespeichert, die stochastisch räumlich organisiert sind und im Gegensatz zu elektronischen Speichermedien keine strukturierte Adressierung erlauben. Daher ist es notwendig, die Adresse selbst in die in einem Strang gespeicherten Daten einzubetten. Auf diese Weise kann man nach der Sequenzierung den ursprünglichen Datenwert wieder zusammensetzen.
Obwohl DNA viele Eigenschaften hat, die sie von bestehenden Speichermedien unterscheiden, gibt es Parallelen zur klassischen Speicherung. Auf der untersten Ebene speichern traditionelle Speichermedien in Raw Bits. Die Speicherlösung abstrahiert die physikalischen Medien, die ein magnetischer Zustand sein können oder die Ladung in einem Kondensator und präsentiert der Speicherhierarchie digitale Rohdaten. In ähnlicher Weise ist die Abstraktion der DNA-Speicherung das Nukleotid: Obwohl ein Nukleotid ein organisches Molekül ist, das aus einer Base (A, C, G oder T) und einem Zucker-Phosphat-Grundgerüst besteht, erfolgt die Abstraktion der DNA-Speicherung als zusammenhängende Folge von quartären (Base-4) Zahlen. *Quelle: Microsoft Research mit University of Washington. (2)

Abb. 1: How_Catalog_encodes_information (Bildquelle: Catalog Infinite Data Archives)
Die von CATALOG entwickelte Methode verfolgt nach Informationen von Cambrigde Consultants einen völlig anderen Ansatz als das traditionelle Denken über die Speicherung von DNA-Daten. In einer Analogie dazu stellen die zu speichernden Informationen ein Buch dar, das durch Kopieren gespeichert werden kann... Eine Übersicht mit weiteren Details finden Sie als angemeldeter Benutzer dieser Webseite in unserer Rubrik Downloads.
Nachdem CATALOG bereits seine selbstentwickelte Methode zur Kodierung von Daten in DNA bewiesen hat, wurde Cambridge Consultants beauftragt, eine Maschine zu bauen, um Daten mit einer Geschwindigkeit von einem Terabit (Tb) in 24 Stunden zu kodieren (entspricht rund 64 Stunden 1080p HD-Video). Solche Geschwindigkeitssprünge sollen laut Entwickler dazu beitragen, dass es wirtschaftlich attraktiver wird, DNA als Medium für die Langzeitarchivierung von Daten zu nutzen. Cloud-Rechenzentren nehmen schon bis zu ein Hektar Land ein und verbrauchen Hunderte von Megawatt an Energie für die Kühlung. Durch die Kodierung von Daten in DNA sollen die Daten mit einem Bruchteil des Energieverbrauchs von Rechenzentren gespeichert werden können. Die DNA-Lagerung kann sicher bei Raumtemperatur geschehen, ist platzsparend und hat laut Catalog eine Lebensdauer von bis zu 1.000 Jahren - wenn sie an einem kühlen und trockenen Ort geschieht. Ohne Leistungsbedarf soll keine aktive Kühlung erforderlich sein.
Der Bau einer Maschine, die Informationen mit solchen Geschwindigkeiten in DNA kodieren kann, ist laut den Entwicklern eine komplexe, multidisziplinäre Herausforderung. Um 1 TB in 24 Stunden zu erreichen, muss die Maschine Dutzende von Milliarden Operationen im mikroskopischen Maßstab und hochparallel mit sehr hoher Genauigkeit durchführen. Cambridge Consultants hat nach eigenen Angaben hierzu ein Projektteam mit einer einzigartigen Kombination von Expertise in den Bereichen Mikrofluidik, Prozessautomatisierung, Softwareentwicklung, Maschinendesign und Synthetische Biologie zusammengestellt.

Abb. 2: Cost to store 1 Petabyte (Bildquelle: Catalog - Infinite Data Archives)
Kommentar Hyunjun Park, Mitbegründer und CEO von CATALOG Technologies: "Um DNA-Datenspeicherung kommerziell nutzbar zu machen, bedarf es erheblicher Fortschritte in der Skalierbarkeit - es ist einfach zu langsam und teuer, um sie in ihrer jetzigen Form für Geschäfts- und Regierungsanwendungen zu nutzen. Die Maschine, die wir mit Cambridge Consultants entwickeln, wird zum ersten Mal in der Geschichte DNA-Datenspeicher aus dem Forschungslabor in die Praxis bringen."
Richard Hammond, Head of Synthetic Biology, Cambridge Consultants, ergänzt: "Wir freuen uns sehr, mit CATALOG an diesem faszinierenden Projekt mit weltveränderndem Potenzial zusammenzuarbeiten. Dieses Projekt erfordert den Aufbau eines sehr heterogenen Teams und stützt sich auf unsere langjährige Erfahrung bei der Durchführung technisch anspruchsvoller Projekte. Es zeigt auch unsere Fähigkeit, Technik und Biologie zu vereinen, um radikal neue Ideen zu verwirklichen."
Quellenangaben mit DNA Storage Links und weiterführenden Informationen:
(1) CATALOG wurde von zwei MIT-Wissenschaftlern gegründet und ist nach eigenen Angaben das erste Unternehmen, das eine Lösung entwickelt hat, um die Speicherung von DNA-Daten kommerziell nutzbar zu machen. Das Unternehmen hat seinen Sitz in Boston, MA.
(2) Microsoft Research DNA Storage, Emerging Storage and Computation Team
- Seit 2015 arbeiten Forscher von Microsoft und der University of Washington zusammen, um DNA als hochdichtes, langlebiges und einfach zu handhabendes Speichermedium zu nutzen. Literaturhinweis: "Random access in large-scale DNA data storage" - Lee Organick, Siena Dumas Ang, Yuan-Jyue Chen, Randolph Lopez, Sergey Yekhanin, Konstantin Makarychev, Miklos Z Racz, Govinda Kamath, Parikshit Gopalan, Bichlien Nguyen, Christopher N Takahashi, Sharon Newman, Hsing-Yeh Parker, Cyrus Rashtchian, Kendall Stewart, Gagan Gupta, Robert Carlson, John Mulligan, Douglas Carmean, Georg Seelig, Luis Ceze, Karin Strauss. Nature Biotechnology | March 2018, Vol 36(3)
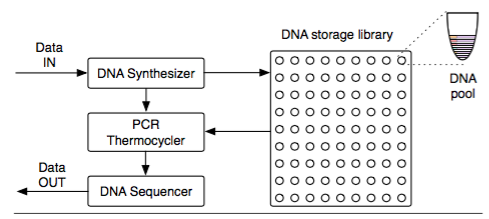
Abb. 3: Schematischer Aufbau eines DNA Storage Archiv Systems (Bildquelle: A DNA-Based Archival Storage System, Figure 3. Overview of a DNA storage system. Research Paper - University of Washington, Microsoft Research, 2016)
Link > Synbiobeta „From magnetic tape to the “DNA hard drive:” entering the next frontier with DNA data storage“
Appendix: Das SRC (Semiconuctor Research Corp.) unterstützt zusammen mit IARPA den Aufbau einer Community zur molekularen Informationsspeicherung im Rahmen des Semiconductor Synthetic Biology for Information Processing and Storage Technologies, (SemiSynBio) Programms. Die Partnerschaft mit der US National Science Foundation und IARPA zielt demnach darauf ab, Entwicklungen im Bereich der Speicherung von DNA-Daten durch die Schnittstelle zwischen synthetischer Biologie und der Halbleiterindustrie zu beschleunigen. Laut Victor Zhirnov, Chief Scientist und Direktor des SRC-Programms sollen mit Programmen wie IARPAs MIST und SRC in den nächsten zehn Jahren wirtschaftlich tragfähige DNA-Datenspeicherlösungen erwartet werden können.
Forschende Unternehmen im Bio-sience Umfeld sind neben den genannten Firmen Microsoft und Catalog derzeit auch Innovatoren wie Twist Bioscience, Evonetix, Molecular Assemblies, Helixworks Technologies, Iridia und Kilobaser (kein Anspruch auf Vollständigkeit).
Querverweis:
*Weiterer Beitrag zu Cambridge Consultants > Das Storage Consortium zu Besuch bei Cambridge Consultants (Sept. 2018) > Siehe hierzu unseren Beitrag "Warum Künstliche Intelligenz neben Hochleistungs-Prozessoren wie GPUs ein schnelles und umfassendes Speichermanagement benötigt".
